Een Y-DNA test. Wat is het en wat heb ik daaraan?
1. DNA testen.
Er komt een moment dat je als Indisch genealogisch onderzoeker tegen een figuurlijke stenen muur aan loopt. Een DNA-test kan dan behulpzaam zijn om de muur te doorbreken. Dit wordt gedaan door het eigen DNA te vergelijken met dat van anderen. Maar let op: er zijn drie soorten DNA die men kan vergelijken. Autosomaal DNA, Mitochondriaal DNA en Y-DNA.
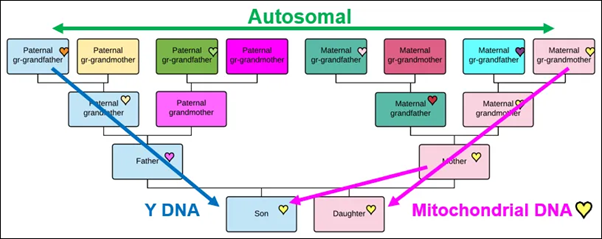
Heeft u zich ooit afgevraagd waarom u uw Y-DNA zou willen testen? Wat zou een Y-DNA test u kunnen vertellen over uw voorouders? Wat zou het voor u betekenen en hoe zou het uw genealogisch onderzoek kunnen helpen?
Als u zoals de meeste genealogen bent, dan wilt u elk detail weten dat u kunt ontdekken over uw voorouders, en Y-DNA vertelt mannen niet alleen over mensen met wie ze nu leven en die voorouders met hen delen op een bepaald moment in de tijd, maar het reikt ook verder terug dan wat genealogie in de traditionele zin ons kan vertellen, voorbij de tijd toen achternamen werden aangenomen, turend in de nevelige sluier van het verleden!
Als u geen man bent, kunt u uw Y-DNA niet rechtstreeks testen, omdat u geen Y-chromosoom hebt. Maar dat hoeft geen probleem te zijn, omdat uw vader of broer of een ander familielid die wel hetzelfde Y-chromosoom draagt als uw vader, misschien wel bereid is om te testen.
Dus laten we het hebben over Y-DNA. In dit artikel leg ik uit wat Y-DNA is, hoe het werkt met matches, of er Indisch Y-DNA is en wat men er uiteindelijk aan heeft.
2. Chromosomen.
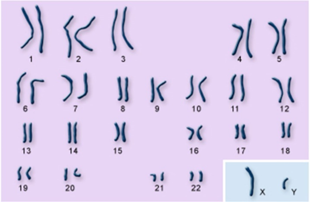
Het 23e paar menselijke chromosomen bestaat uit een X- en een Y-chromosoom. Vrouwelijke kinderen erven een X van beide ouders. Mannelijke kinderen erven een X-chromosoom van hun moeder, maar een Y van hun vader. In het algemeen volgt het Y-chromosoom de mannelijke achternaamlijn. Mannen geven hun Y-chromosoom alleen door aan hun zonen. Bij adopties komt de familienaam natuurlijk niet overeen met de biologische “familienaam” die aan het Y-chromosoom verbonden is. De andere 22 paar chromosomen noemen we autosomen.
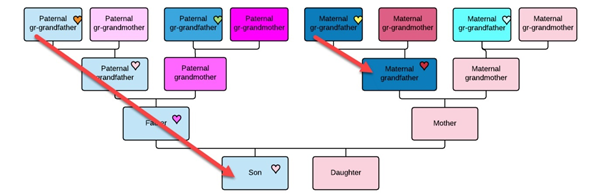
In het voorbeeld hieronder kunt u zien dat het lichtblauwe Y-chromosoom wordt doorgegeven van vader op zoon op zoon op zoon naar het mannelijke kind in de huidige generatie.
De donkerblauwe overgrootvader van moederszijde in dit voorbeeld geeft ook zijn Y-chromosoom door aan zijn zoon, maar daar houdt het op, want de volgende generatie in deze stamboom is een vrouw.
De lichtblauwe zoon onderaan erft een Y-chromosoom van zijn vader, van voorouders helemaal boven in die lichtblauwe lijn, samen met zijn achternaam. De dochter krijgt geen Y-chromosoom.
Bij een Y-DNA test kijken we niet naar hoeveel stukjes DNA wij delen met anderen, zoals bij een autosomaal DNA test. Bij autosomaal DNA drukken we het aantal overeenkomstige stukjes DNA uit in centi Morgan (cM) en we kunnen ook zien waar op welk chromosoom we precies hetzelfde stukje DNA delen met iemand anders. Hoe meer cM en hoe meer stukjes, hoe groter de kans is dat degene waarmee een match wordt gevonden daadwerkelijk familie is. Bij autosomaal DNA krijgt men willekeurige stukjes van beide ouders en die weer op hun beurt van hun ouders etc. Soms is er na een paar generaties helemaal niets herkenbaars meer over van het doorgegeven DNA van een bepaalde voorouder. Een autosomaal DNA test kijkt over het algemeen maximaal een generatie of 8 terug omdat het simpelweg iedere generatie wordt verdund en gemixt.
3. Y-DNA werkt anders.
Bij Y-DNA werkt dat helemaal anders. Omdat het Y-DNA slechts heel weinig muteert (verandert) als het wordt overgedragen van vader op zoon, kan men zien waar de mannelijke voorouders oorspronkelijk vandaan kwamen. Het Y-DNA muteert wel langzaam, maar je moet in de duizenden jaren denken om hierdoor grote verschillen te zien. Als we die verschillen in kaart brengen vanaf de eerste man – laten we die voor het gemak Adam noemen – zien we dat het Y-DNA over vele duizenden jaren zodanig muteert, dat we de mutaties kunnen indelen in zogenaamde “haplogroepen”. Denk aan een haplogroep als een genetische clan die je vertelt tot welke genetische familiegroep je behoort, zowel vandaag als in het verleden.
We kijken bij het Y-DNA dus niet naar stukjes autosomaal DNA die worden doorgegeven van willekeurige voorouders in willekeurige volgorde en formaat. We kijken alleen naar mutaties op het Y-chromosoom.
4. Mutaties.
We zijn blij met mutaties. Als er nooit mutaties zouden zijn opgetreden, zou het Y-DNA van alle mannen identiek zijn en dus niet bruikbaar voor ons voor genealogie of om verder terug te kijken voorbij de tijd waarin achternamen ontstonden.
Y-DNA kan worden getest op twee verschillende soorten mutaties, STR-markers (short tandem repeats) en SNP’s (single nucleotide polymorphisms). Er zijn veel STR-markers en veel SNP’s.
Eerst iets over DNA. DNA bestaat uit basisparen met vier verschillende nucleotiden, afgekort A, C, G en T. A=adenine, C=cytosine, G=guanine en T=thymine. Ze vormen samen de typische dubbele helix.
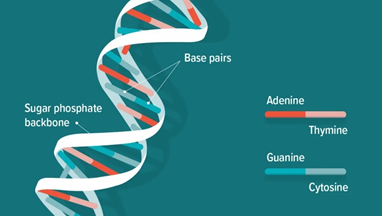
insertie.
Een deletie treedt op wanneer een nucleotide, of meerdere nucleotiden, bij de celdelingsproces niet worden gekopieerd. Door DNA schade of fouten bij het DNA replicatieproces. Die plaats of plaatsen zijn dan blanco.
We gaan uit van het volgende voorbeeldsegment.
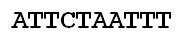
Er zou sprake zijn van een deletie als de leidende A (of een reeks nucleotiden) gewoon zou verdwijnen.
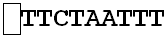
Er is sprake van vervanging wanneer een nucleotide wordt vervangen door of verwisseld met een andere nucleotide. Zo zou bijvoorbeeld een A kunnen worden vervangen door een van de andere nucleotiden zoals G, enzovoort.
Een vervanging zou plaatsvinden als de eerste A in het voorbeeldsegment zou veranderen in T of G of C, zoals in het onderstaande voorbeeld:
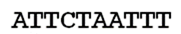
wordt

Deze vervanging is een mutatie. Omdat het een enkele vervanging is, wordt dit een single nucleotide polymorphism genoemd, afgekort SNP.
Een insertie vindt plaats wanneer een nucleotide of een groep nucleotiden wordt gedupliceerd en tussen bestaande nucleotiden wordt ingevoegd.
Van een insertie is sprake wanneer DNA wordt ingevoegd tussen bestaande nucleotideplaatsen.
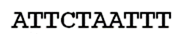
wordt

Let op de extra rode CT’s die zijn ingebracht. Om precies te zijn, 4 extra CT’s, voor een totaal van 4 sets van CT. Dit is de definitie van een zogenaamde STR, een short tandem repeat mutatie.
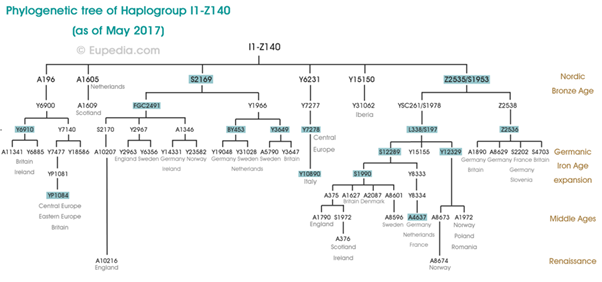
Het komt er dus op neer dat we bij de Y-DNA test een zogenaamde “terminal SNP” kunnen bepalen. Terminale SNP? Nee, het is echt niet terminaal. In deze context betekent “terminaal” einde van de lijn, dus het verst naar beneden en het dichtstbij aanwezig in de haplotree (boom). Bepaalde mutaties komen vaker voor in bepaalde gebieden.
Als men een uitgebreide test doet, kan ook worden geüpload naar een onafhankelijke database YFull. Hieronder de weergaven in YFull die dus feitelijk ook matches weergeeft.
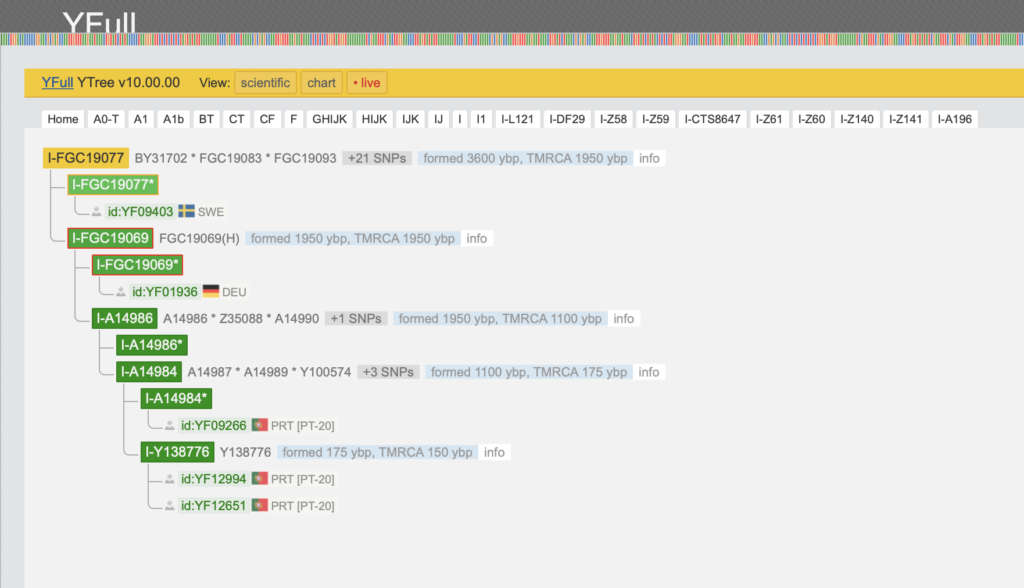
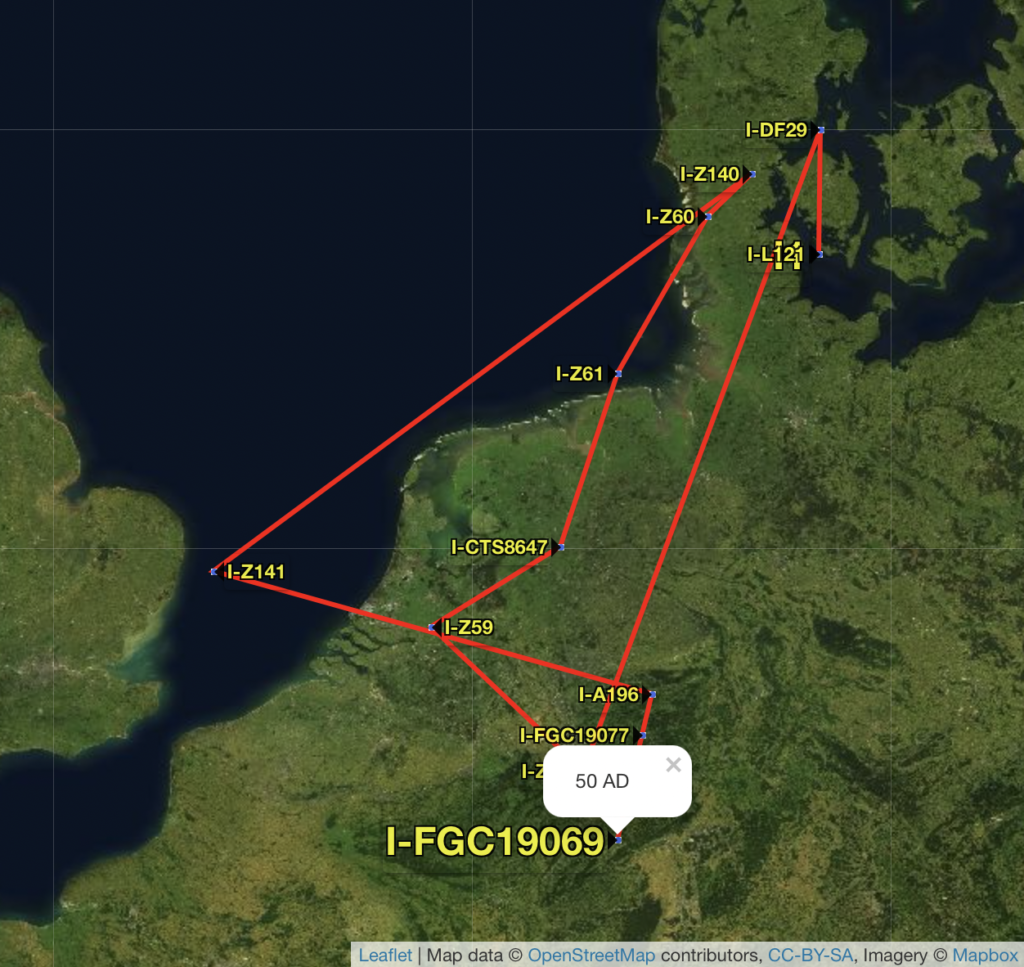
Mijn terminal SNP is I-FGC19069 (FGC is Full Genomes Corporation laboratorium) die ook andere namen heeft zoals FGC113 en BY31702. De SNP is ontstaan 1950 “years before present”. Bij DNA onderzoek wordt er dus niet gesproken van “BC” (before Christ) maar van YBP. U ziet dat er nog iemand uit Zweden in de tak er boven FGC19077 zit. Ik ben daar van afgesplitst en zit nu in mijn eigen mini-groep. Bij mij staat er DEU omdat mijn voorvader vermoedelijk uit Duitsland kwam.

6. STR’s.
STR’s, dus korte tandem herhalingen, vertonen wat lijkt op kopieermachinefouten. Dit gebeurt wanneer een specifiek segment van het Y-DNA verschillende keren na elkaar wordt herhaald. Dus een groep ingevoegde nucleotiden die allemaal duplicaten van elkaar zijn.
In het voorbeeld in paragraaf 4 hebben we een korte tandem-herhaling die 4 segmenten lang is, wat betekent dat CT 4 keer is ingevoegd. Met andere woorden, als dit marker DYS390 is, heb je een waarde van 5, wat 5 herhalingen van CT betekent
De mutaties kunnen zich voordoen tussen een kleinzoon en zijn grootvader, of er kunnen zich honderden of mogelijk zelfs duizenden jaren geen mutaties voordoen op een marker. Dat is voor iedere marker anders.
SNP’s worden beschouwd als “eens in je leven”, of misschien beter gezegd, “eens in het leven van de mensheid”, terwijl STR’s “de hele tijd” voorkomen in elke haplogroep.
Dus STR-markers worden gebruikt voor genealogische matching, terwijl SNP-markers samenwerken met STR-markers om de genealogie verder te verfijnen, plus een gedetailleerde haplogroep opleveren.
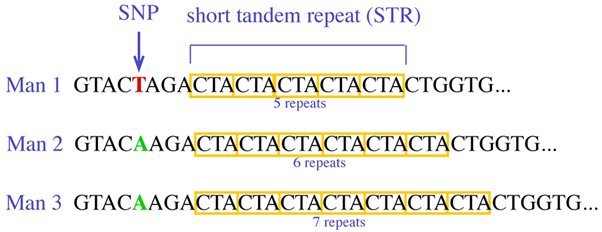
STR’s worden dus ingedeeld in markers. In het plaatje hieronder zijn de eerste 12 markers zichtbaar. Een marker heeft een naam, bijvoorbeeld DYS393 of DYS392. Elke marker heeft een waarde. De waarde kan veranderen door een STR, een short tandem repeat mutatie.
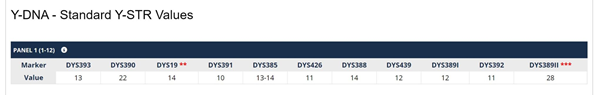
Een marker springt dan van bijvoorbeeld 12 naar 13. Of terug van 15 naar 14. De getallen heten formeel allelwaarden. Als je twee broers zou vergelijken of een vader en een zoon dan is de kans 99,9 % dat alle gemeten allelwaarden gelijk zijn. Maar een verre neef kan al een klein verschil hebben. Hoe meer verschillen hoe verder weg de gezamenlijke voorvader ligt.
Hieronder de vergelijking tussen mijn eigen allelwaarden en die van iemand anders:

STR’s zijn nuttig voor genealogische doeleinden, om te bepalen met wie je overeenkomt binnen een recent tijdsbestek van, zeg, de afgelopen 500 jaar of zo.
De verschillen in allelwaarden op de STR-markers bepalen hoe lang geleden men een gezamenlijke voorouder had. Hoe minder mutaties, dus dezelfde waarde per marker, hoe dichterbij de voorouder is in de tijd.
Ondanks dat men dus met iemand een terminal SNP deelt, en dus in dezelfde tak van een haplogroep zit (in mijn geval hierboven is dat I-A5475 dat precies 1 niveau boven mijn terminal SNP ligt), is er toch een groot aantal STR’s dat verschillende waarden heeft.
7. STR matches
Het bedrijf waar getest wordt, in dit geval Family Tree DNA, zet in een overzicht welke mannen het dichtst in de buurt komen, dus minder verschillen in allelwaarden hebben. Dat ziet er dan zo uit.
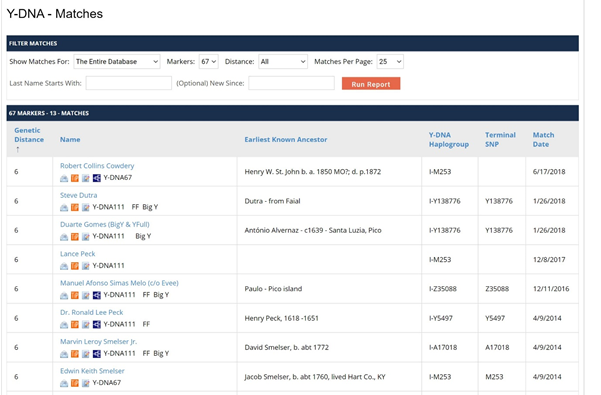
Dit een zogenaamde “Y-67 test”. Er worden hier dus slechts 67 STR markers getest. Men hoopt andere testers te vinden die ook hebben getest en die zo min mogelijk verschillen in waarden in hun markers hebben ten opzichte van het eigen Y-DNA. Dit zijn dus de Y-DNA matches. De “genetic distance” is hier 6. Dat zijn geen generaties maar “genetische afstanden”.
Wanneer we het hebben over twee of meer STR’s is genetische afstand het totale aantal verschillen of mutaties tussen twee reeksen resultaten. In het algemeen wordt dit gevonden door de verschillen tussen elke STR-marker op te tellen.
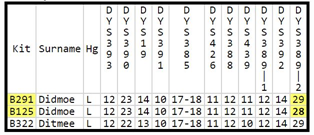
Bijvoorbeeld, set B291 en B125 hebben allelwaarden van respectievelijk 29 en 28 op marker DYS389II. Dit is een verschil van 1 (29-28= 1). Omdat dit het enige verschil is in hun Y-DNA12-profielen (haplotypen), is hun genetische afstand 1.
In dit voorbeeld hebben de kits B291 en B322 verschillen bij DYS390, DYS19, en DYS439. Het verschil voor elk wordt berekend (DYS390: 23-22= 1, DYS19: 14-13= 1, DYS439: 11-10= 1). De verschillen worden bij elkaar opgeteld. Het totale aantal verschillen is 3 (1+1+1= 3). Hun genetische afstand op 12 STR-markers is dan 3.
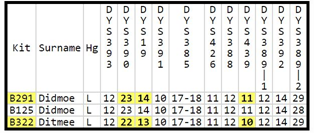
Maar let op! in dit voorbeeld is het dus de genetische afstand voor een groep van slechts 12 markers. Als men 700 markers test, kan de genetische afstand dus vele malen groter zijn. Hoe kleiner de genetische afstand, hoe minder ver in het verleden er een gezamenlijke voorvader was.
Het is raadzaam om deel te nemen aan een project dat hoort bij de gevonden haplogroep. De moderator van de groep sorteert de deelnemers op basis van meerdere factoren zodat er een groep wordt gevormd van mensen die het dichtst bij elkaar zitten.
Een overzicht ziet er dan zo uit. Dit is slechts een klein deel.

8. Sociale media.
Facebook is voor iedereen, die zich met DNA onderzoek in samenhang met genealogie bezighoudt, een uitstekend medium om kennis te vergroten en informatie te delen. Er zijn FB groepen die specifiek voor een bepaalde haplogroep zijn, maar ook veel FB groepen die algemeen DNA testen bespreken of FB groepen voor DNA technisch gevorderden.
9. Conclusie.
Ik heb tot nu toe uitgelegd wat Y-DNA is, wat STR’s en SNP’s zijn, hoe matches worden bepaald. Terug naar de vraag: Een Y-DNA test? Wat heb ik er aan?
Omdat Y-DNA rechtstreeks geërfd wordt van de patrilineaire (mannelijke) lijn, in tegenstelling tot autosomaal DNA, is er geen genetische bijdrage van de vrouwelijke voorouders. Deze unieke mannelijke overerving stelt ons in staat Y-DNA te gebruiken om andere mannen te matchen, te beginnen met de eerste generatie, de vader, en dan vele generaties terug, waardoor we ons voorouderlijk erfgoed kunnen bekijken voorbij de lijn-in-het-zand grens met achternamen.
Sommige mensen, vooral nieuwe autosomale testers, geloven dat Y-DNA alleen nuttig is voor wat we noemen “deep ancestry”, in het Nederlands vertaald als “verre voorouders” en niet voor genealogie. Dat is onjuist. Y-DNA is erg belangrijk voor het bevestigen van de afstamming van bekende voorouders. In feite, zonder Y-DNA, kan je met alleen autosomale testen het verschil niet zien tussen de vaderskant en de moederkant.
Y-DNA testen kunnen helpen om er achter te komen waar de mannelijke voorouder vandaan kwam. Bijvoorbeeld als een man bij zijn huwelijk aangeeft dat de vader onbekend is. Zoals in mijn geval:

Met een Y-DNA test heb ik kunnen vaststellen dat de onbekende vader zijn oorsprong had in Noord-West Europa (Haplogroep I) en niet in Azië (Haplogroep O). De onbekende vader van mijn overgrootvader was dus een Europeaan. Door een nazaat van de broer van de moeder te testen, weet ik dat er waarschijnlijk ook geen sprake is geweest van incest, dus dat de moeder niet zwanger is gemaakt door de vader of de broer, omdat de nazaat van de broer een volledig andere haplogroep heeft (R). Dus met een Y-DNA test kunnen verbanden worden uitgesloten of bewezen.
Nog een ander voorbeeld. Iemand waarmee ik een kleine autosmaal DNA match heb, vertelt het verhaal dat het gerucht gaat dat haar grootvader een kind is van een pastoor. Er zijn vermoedens welke pastoor het betreft en zijn familie is met publicaties en bronnen opgespoord. De kleinzoon van de broer van de pastoor wil een Y-DNA test doen. De broer van de dame ook. Als het resultaat overeenkomt, dan weten we dat de pastoor de vader is.
Geadopteerde mannen gebruiken een Y-DNA test om hun biologische vader te vinden. Als blijkt dat in de database er een genetische afstand van 0 is en de terminal SNP komt overeen dan is die persoon in directe mannelijke lijn verbonden aan de geadopteerde. En daarmee wordt het vinden van de biologische vader plotseling in een stroomversnelling gebracht.
Y-DNA kan ook behulpzaam zijn als er sprake is van patroniemen. Een patroniem of vadersnaam is een naam, al dan niet officieel, die aangeeft hoe de vader van de naamdrager heet. Voordat de achternaam officieel werd ingevoerd werd een kind vaak in een doopboek ingeschreven met de voornaam en de ouders werden ook niet altijd met de achternaam genoemd. Simpelweg omdat er nog geen officiële achternamen bestonden. Dus als er in een doopakte staat: “Gedoopt Claes zoon van Claes Jans en Trijntje” dan wordt het lastig om te bevestigen welke Jan dan de vader was van Claes. Als men een vermoeden heeft welke man een nazaat is van Jan, kan een Y-DNA test uitkomst bieden. Zo ook bij erkenningen in Nederlands Indië. Als er een vermoeden bestaat wie het kind erkend heeft en er is met zekerheid een mannelijke nazaat te vinden, dan kan een Y-DNA test ook hier uitkomst bieden.
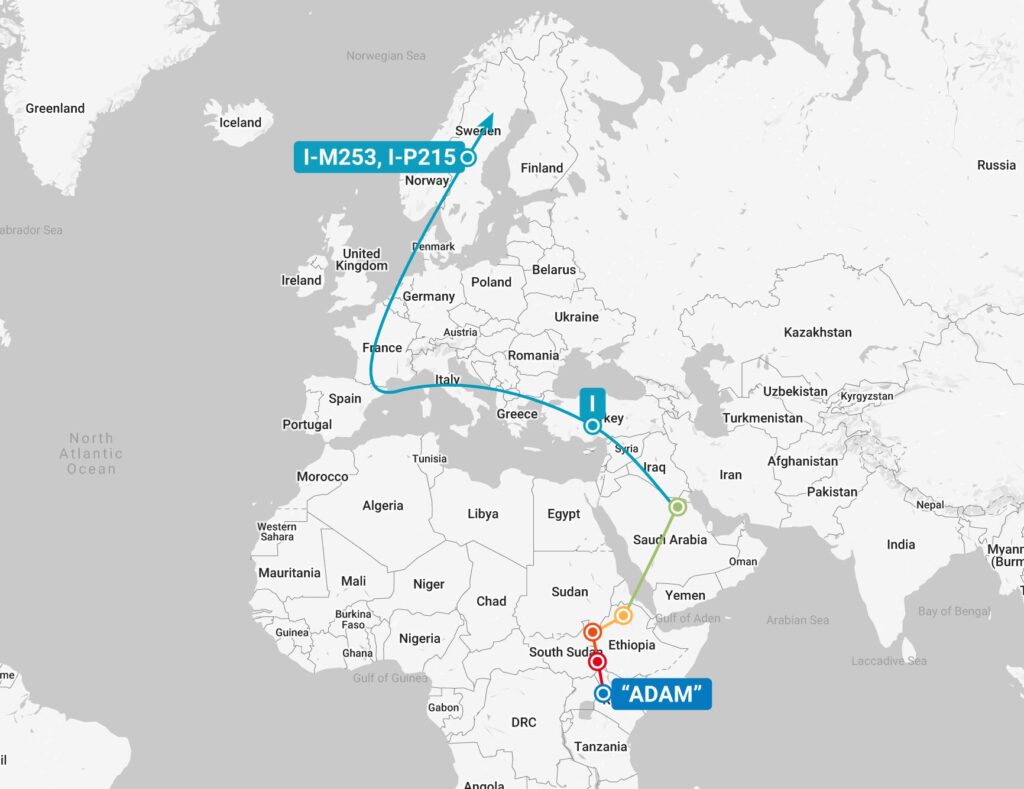
Y-DNA-testen zijn ook de beste manier om de genealogie verder terug in de tijd te brengen. Door middel van overeenkomsten met andere testers en haplogroep-overeenkomsten kan er iets gezegd worden over de oorsprong van de mannelijke voorvaderen. Te beginnen met Y-lijn Adam, lang geleden geboren in Afrika, tot waar we nu zijn. Dit zijn zaken die autosomaal DNA eenvoudigweg niet kan bewerkstelligen.
Er zijn ook leuke tools beschikbaar. In onderstaand plaatje wordt geplot waar de mannelijke voorouders mogelijk hebben gewoond en zijn verhuisd op basis van hun SNP Y-DNA mutaties. Voorlopig wordt er dus 1950 jaar geleden gepinpoint in Duitsland, komende vanuit de Deense eilanden.
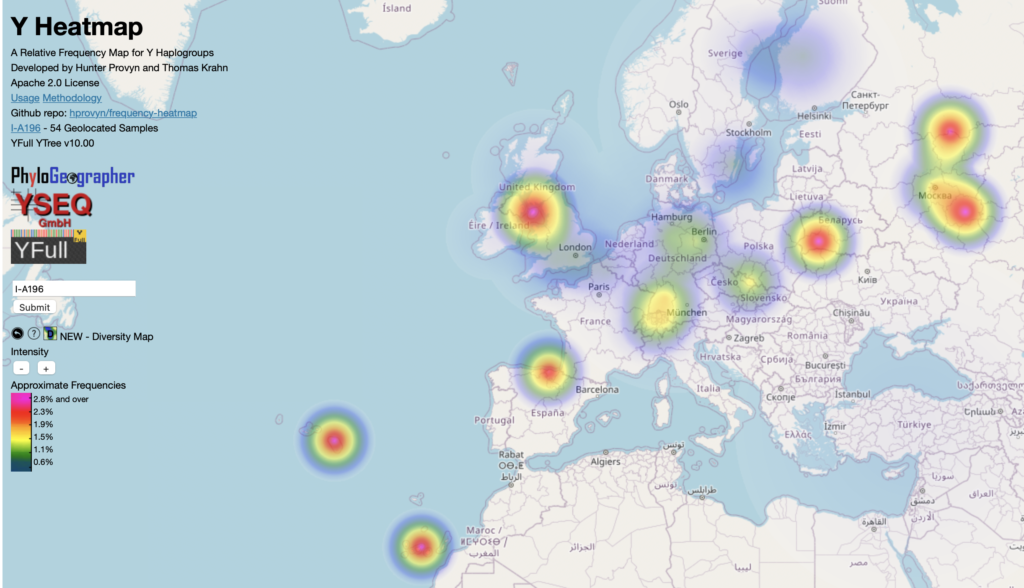
Y-Heatmap SNP A196, 2 takken boven mijn terminal SNP.